![图片[1] - AI科研 编程 读书笔记 - 【人工智能】PlantAIM: 一种融合全局注意力与局部特征以提升植物病害识别能力的新型基准模型 - AI科研 编程 读书笔记 - 小竹の笔记本](https://img.smallbamboo.cn/i/2025/04/10/67f7abfee6753.png)
![图片[2] - AI科研 编程 读书笔记 - 【人工智能】PlantAIM: 一种融合全局注意力与局部特征以提升植物病害识别能力的新型基准模型 - AI科研 编程 读书笔记 - 小竹の笔记本](https://img.smallbamboo.cn/i/2025/04/10/67f7abcae5045.png)
GitHub:https://github.com/abelchai/PlantAIM
文章在2025年1月31日被接收。
作者团队:
斯威本科技大学砂拉越校区,马来西亚:Abel Yu Hao Chai、Sue Han Lee、Fei Siang Tay
法国蒙彼利埃大学:Hervé Goëau、Pierre Bonnet
法国国家信息与自动化研究所,蒙彼利埃:Alexis Joly
动机
- 问题背景:植物病害严重影响农业产量与质量,传统依赖病理学专家与实验室的方法成本高、耗时长,难以快速响应病害爆发。
- 技术挑战:现有深度学习方法(如ViT和CNN)在多作物病害识别任务中表现不足,需同时学习作物特异性特征(如叶脉形态)与病害特异性特征(如不规则病斑),但两类特征提取机制差异大,现有模型缺乏有效融合策略。
- 核心目标:提出PlantAIM模型,通过整合ViT的全局注意力机制(捕捉病害长程依赖)与CNN的局部空间特征提取能力,提升多作物病害识别性能,并探索模型在真实环境与有限样本下的鲁棒性。
核心方法
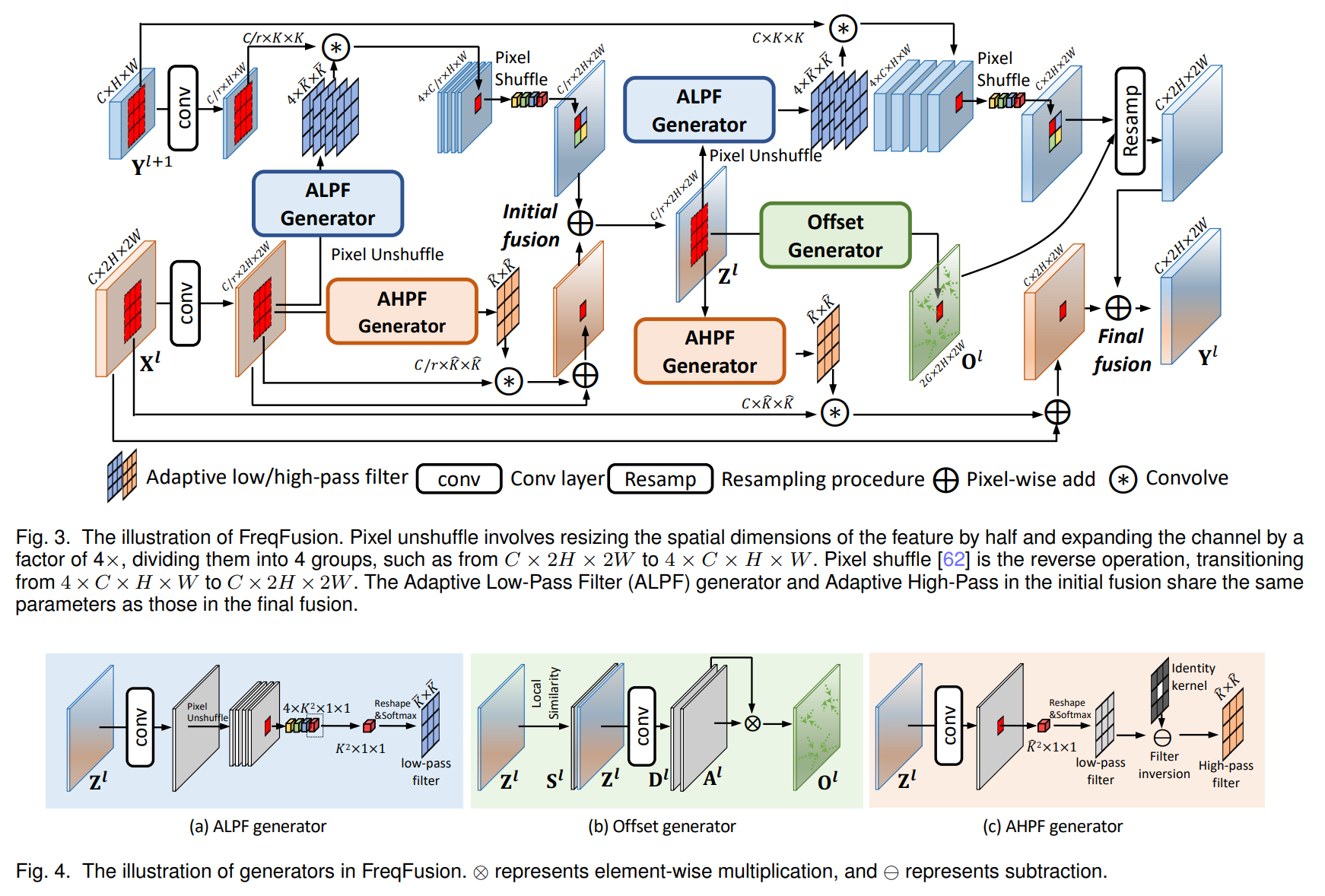
- 模型架构:
- 双骨干网络:采用预训练的ViT(提取全局特征)和ResNet152(提取局部特征),通过MLP对齐特征维度。
- 特征融合策略:
- 全局-局部特征融合:将ViT的CLS token(全局特征)与CNN特征通过乘法融合(优于加法),增强特征交互。
- GLFA层(Global-Local Fusion Attention):通过自注意力机制动态融合全局特征与ViT的局部图像块特征,生成更具判别力的综合特征。
- 双分类头:独立预测作物种类(14类)与病害类型(21类),仅当两者均正确时判定为正确识别,提升任务解耦能力。
- 训练策略:
- 损失函数:作物与病害分类任务分别计算交叉熵损失((L_p)和(L_d)),联合优化。
- 残差连接:保留ViT CLS token特征,缓解特征遗忘问题。
![图片[3] - AI科研 编程 读书笔记 - 【人工智能】PlantAIM: 一种融合全局注意力与局部特征以提升植物病害识别能力的新型基准模型 - AI科研 编程 读书笔记 - 小竹の笔记本](https://img.smallbamboo.cn/i/2025/04/10/67f7ad2489579.png)
实验结果
- 性能对比(SOTA模型):
- PV数据集(实验室环境):PlantAIM(1H单分类头)达到99.66%准确率,超越现有最佳模型(98.86%)0.8%。
- 有限样本场景(PV limited):在仅10样本/类的情况下,PlantAIM(2H双分类头)准确率64.92%,显著优于ViT(59.19%)和CNN(55.61%)。
- 真实环境数据集:在IPM、Bing、PlantDoc上,PlantAIM分别取得**42.74%、42.86%、38.85%**准确率,优于ViT和CNN模型。
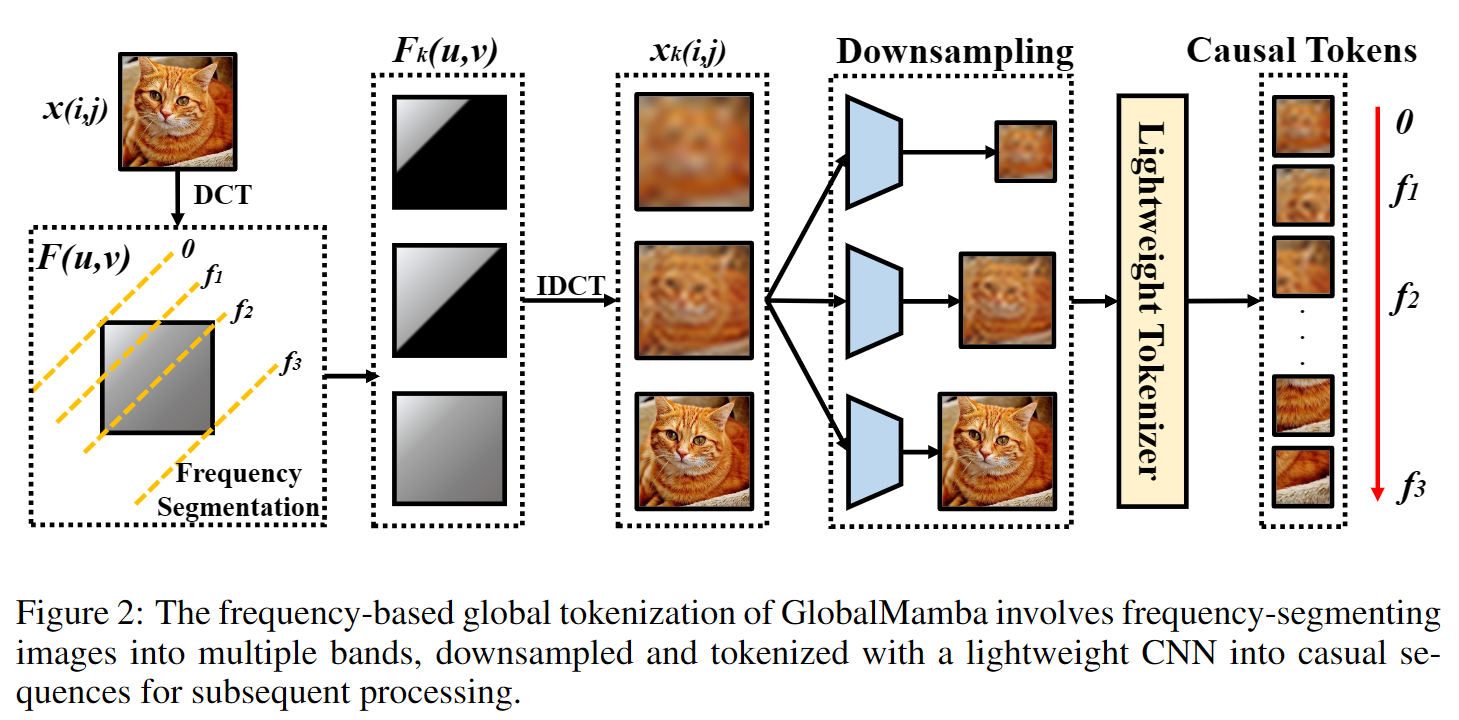
- 可视化分析:
- Grad-CAM热力图显示,CNN关注叶片形态(如叶脉),ViT聚焦病斑区域,而PlantAIM能同时捕获两类特征(例如苹果黑腐病中,作物分类关注叶缘,病害分类聚焦内部病斑)。
- 特征分布分析:PlantAIM学习到的特征与分类器权重距离更大,表明其特征更具判别力且避免对单一特征的过度依赖。
对比算法
- 基线模型:
- CNN模型:ResNet152、DenseNet121、InceptionV3等,在PV数据集上最高99.48%准确率。
- ViT模型:ViT-base在PV上达99.57%,但真实环境下泛化能力受限。
- 混合模型:如[42](CNN+注意力)、[50](ViT+CNN层级结构),最高98.86%准确率。
- PlantAIM优势:通过GLFA层实现动态特征融合,平衡全局与局部信息,显著提升跨域泛化能力。
数据集
- 训练集:Plant Village(PV)数据集,54,305张实验室图像,涵盖14种作物、21种病害,38种作物-病害组合。
- 数据划分:80%训练,20%测试,其中Apple black rot等3类病害各仅10样本用于模拟小样本场景。
- 测试集:
- PV seen:常规测试集(20%)。
- PV limited:3类小样本病害测试集。
- 真实环境数据:IPM(复杂背景)、Bing(网络爬取图像)、PlantDoc(17种病害),用于评估模型泛化性。
![图片[4] - AI科研 编程 读书笔记 - 【人工智能】PlantAIM: 一种融合全局注意力与局部特征以提升植物病害识别能力的新型基准模型 - AI科研 编程 读书笔记 - 小竹の笔记本](https://img.smallbamboo.cn/i/2025/04/10/67f7ad5d383a7.png)
改进空间
- 极端小样本场景:在仅1-5样本/类的情况下,模型性能仍有下降,需探索元学习或数据增强策略。
- 计算效率:PlantAIM依赖双骨干网络,推理时间(1.58分钟/epoch)高于单模型(ViT为1.06分钟),未来可优化轻量化设计。
- 多模态融合:结合分子检测或环境传感器数据,提升复杂病害(如病毒病)的识别精度。
- 长尾分布问题:PV数据集中部分病害样本稀缺,需引入重加权损失或对比学习优化类别平衡。
总结
PlantAIM通过创新性的全局-局部特征融合机制,在多作物病害识别任务中树立新基准,并为复杂农业场景下的病害检测提供了可扩展框架。未来工作可聚焦小样本学习与模型轻量化,进一步推动农业智能化应用。
© 版权声明
1. 除特殊说明外,本网站所有原创文章的版权归作者所有,未经授权,禁止以任何形式(包括但不限于转载、摘编、复制、镜像等)发布至任何平台。
2. 论文总结类文章中涉及的图表、数据等素材,版权归原出版商及论文作者所有,仅为学术交流目的引用;若相关权利人认为存在侵权,请联系本网站删除,联系方式:i@smallbamboo.cn。
3. 违反上述声明者,将依法追究其相关法律责任。
2. 论文总结类文章中涉及的图表、数据等素材,版权归原出版商及论文作者所有,仅为学术交流目的引用;若相关权利人认为存在侵权,请联系本网站删除,联系方式:i@smallbamboo.cn。
3. 违反上述声明者,将依法追究其相关法律责任。
THE END




























暂无评论内容